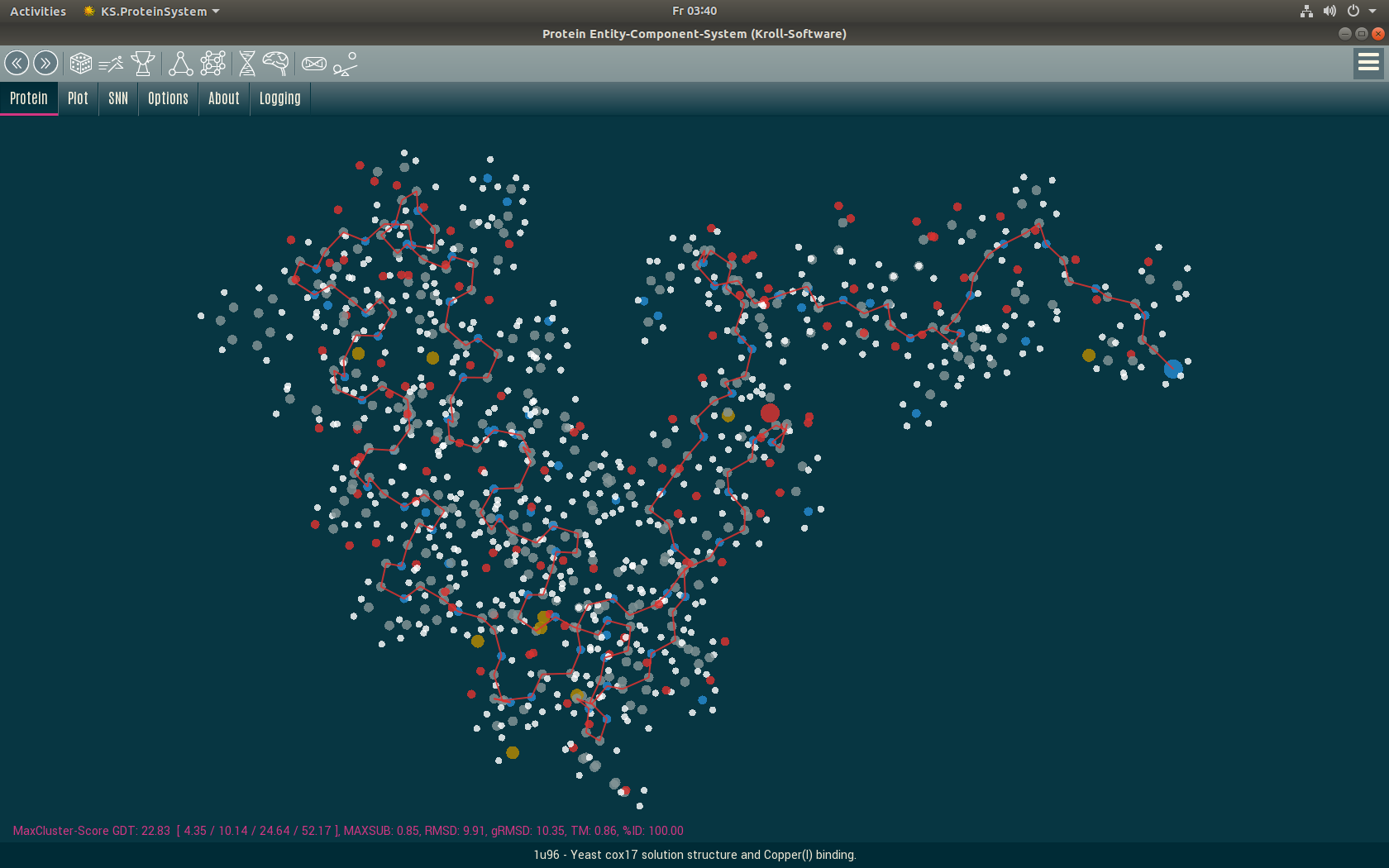
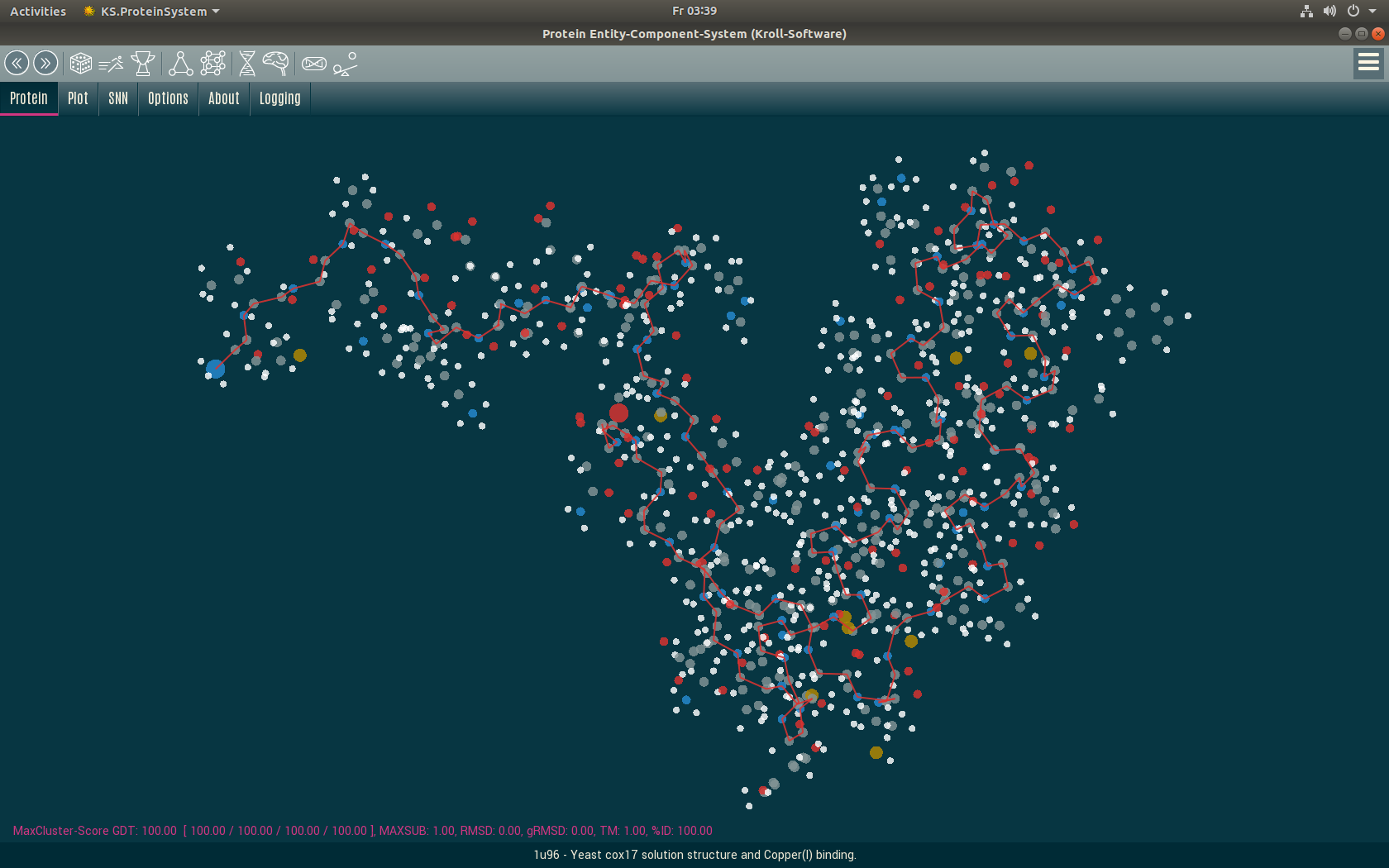
Chiralität in der Proteinfaltung
In der Proteinfaltung gibt es zu jedem Protein auch eine gespiegelte Form. Bisher ist es schwierig zu sagen, ob ein Protein in der gespiegelten Form vorliegt.
In diesem Artikel beschreiben wir einen Weg, wie die Chiralität eines Proteins durch ein Neuronales Netzwerk mit Tensorflow bestimmt werden kann.
Unser Verfahren
Das gesamte Verfahren wird hier vollständig beschrieben, damit jeder es nachmachen und weiter verbessern kann. Dieses Verfahren ist eine vollständig eigene Entwicklung von Kroll-Software und basiert auf keinen anderen Ideen. Bitte zitieren sie uns, wenn Sie dieses Verfahren verwenden.
Das Verfahren macht eine Voraussage über die Chiralität eines Proteins.
Motivation
Wenn ein Protein durch einen Algorithmus gefaltet wird, kann es oft geschehen, dass es falsch herum gefaltet wird. Daher ist es wichtig, seine Chiralität zu erkennen, um es zu spiegeln, wenn dies erforderlich ist.
Daten
Die verwendeten Trainings-Daten stammen aus der öffentlich zugänglichen Protein Datenbank (PDB).
Implementierung
Das Neuronale Netzwerk nutzt Keras / Tensorflow (oder ein anderes Backend). Wir programmieren in C# und verwenden eine modifizierte Version von Keras.NET. Alle Codebeispiele sind daher in C#, lassen sich aber leicht in andere Sprachen adaptieren.
Netzwerk Architektur / Topologie
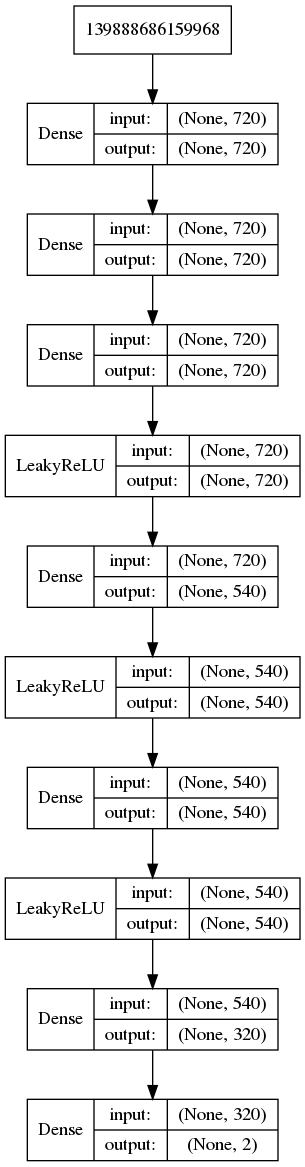
BaseModel BuildModel()
{
bool useBias = false;
string initializer = "random_uniform";
Sequential model = new Sequential();
model.Add(new Dense(units: 720, input_dim: 720));
model.Add(new Dense(units: 720, activation: "tanh", use_bias: useBias, kernel_initializer: initializer));
model.Add(new Dense(units: 720, activation: "tanh", use_bias: useBias, kernel_initializer: initializer));
model.Add(new LeakyReLU());
model.Add(new Dense(units: 540, use_bias: useBias, kernel_initializer: initializer));
model.Add(new LeakyReLU());
model.Add(new Dense(units: 540, use_bias: useBias, kernel_initializer: initializer));
model.Add(new LeakyReLU());
model.Add(new Dense(units: 320, use_bias: useBias, kernel_initializer: initializer));
model.Add(new Dense(units: 2, activation: "softmax"));
var optimizer = new Adam(lr: 0.25e-5f);
model.Compile(optimizer: optimizer,
loss: "categorical_crossentropy",
metrics: new string[] { "accuracy" });
model.Summary();
model.Plot("chirality-model.png");
return model;
}
Eingabewerte
Das Modell erhält alle Winkel Phi und Psi der Alphakette des Proteins in einem vektorisierten Array.
Vektorisierungs-Funktion
float[] Vectorize(double[] Phi, double[] Psi, bool randomize = false)
{
float[] arr = new float[720];
foreach (double d in Phi)
{
double degree = d.ToDegree();
int idx = (int)(degree + 180.5);
while (idx < 0)
idx += 360;
while (idx >= 360)
idx -= 360;
if (randomize)
RandomizeIndex(idx);
arr[idx] += 1;
}
foreach (double d in Psi)
{
double degree = d.ToDegree();
int idx = (int)(degree + 180.5);
while (idx < 0)
idx += 360;
while (idx >= 360)
idx -= 360;
if (randomize)
RandomizeIndex(idx);
arr[idx + 360] += 1;
}
if (randomize)
AddNoise(arr, Phi.Length / 10);
// Normalize
float max = arr.Max();
for (int j = 0; j < 720; j++)
arr[j] /= max;
return arr;
}
Randomisierung
Wir randomisieren den Eingabevektor für grössere Robustheit.
int RandomizeIndex(int idx)
{
const int range = 45;
int r = ThreadSafeRandom.Next(range);
if (ThreadSafeRandom.NextDouble() < 0.5)
r = -r;
int ret = idx + r;
while (ret < 0)
ret += 360;
while (ret >= 360)
ret -= 360;
return ret;
}
Rauschen
Wir fügen auch etwas Rauschen hinzu.
float[] AddNoise(float[] vec, int count)
{
int half = vec.Length / 2;
for (int i = 0; i < count; i++)
{
int idx = ThreadSafeRandom.Next(360);
vec[idx] += 1;
}
for (int i = 0; i < count; i++)
{
int idx = ThreadSafeRandom.Next(360);
vec[idx + half] += 1;
}
return vec;
}
Training
Dies ist unsere Trainingsfunktion in Keras.NET / C#
public double[] BatchTrain(List data, bool evaluate = false, bool randomize = false)
{
int batch_size = 32;
int batchStart = 0;
if (evaluate)
batch_size = data.Count;
List retLoss = new List();
List retErr = new List();
MRE.Reset();
var ts = PythonEngine.BeginAllowThreads();
try
{
while (batchStart < data.Count)
{
int count = Math.Min(batch_size, data.Count - batchStart);
float[,] chirlist = new float[count, 720];
float[,] yval = new float[count, 2];
for (int j = 0; j < count; j++)
{
ChiralityData chir = data[j + batchStart];
float[] vec = Vectorize(chir.Phi, chir.Psi, randomize);
for (int k = 0; k < 720; k++)
{
chirlist[j, k] = vec[k];
}
yval[j, 0] = chir.Mirrored ? 0 : 1;
yval[j, 1] = chir.Mirrored ? 1 : 0;
}
using (Py.GIL())
{
var train = new NDarray(chirlist);
var y = new NDarray(yval);
double[] retVal = null;
if (evaluate)
retVal = Model.Evaluate(train, y, verbose: 0);
else
retVal = Model.TrainOnBatch(train, y);
retLoss.Add(retVal[0]);
retErr.Add(retVal[1]);
}
batchStart += batch_size;
}
}
catch (Exception ex)
{
ex.LogError();
HasErrors = true;
}
finally
{
PythonEngine.EndAllowThreads(ts);
MRE.Set();
}
double lossVal = retLoss.Sum() / retLoss.Count;
double errVal = retErr.Sum() / retErr.Count;
return new double[] { lossVal, errVal };
}
Credits
Citation needed.
Unterstützen Sie diese Forschung
Wenn Ihnen diese Forschung gefällt, freuen wir uns auf Ihre Unterstützung. Bitte kontaktieren Sie uns.